Biologie Moléculaire :
Chapitre 2 : les Biotechnologies
Partie 3 : Les outils du génie génétique.
Télécharger le document (chapitre 2 entier) sur le serveur FTP ou sur le serveur web (HTTP).
I Les enzymes.
On va se servir des enzymes pour couper, coller et synthétiser des acides nucléiques.
A Les polymérases.
Toutes les polymérases agissent de 5’ vers 3’. En cas d’erreur, il faut réparer de 5’ vers 3’ pour permettre la correction et la re-synthèse.
1 Les DNA polymérases.
a Les DNA polymérases DNA dépendantes.
Ces DNA polymérases fabriquent de l’ADN à partir d’une matrice d’ADN. On trouve trois activités chez ces enzymes :
- Synthèse de 5’ vers 3’ à partir d’une amorce hybridée (matrice simple brin hybridée)
- Activité 3’5’ exonucléase.
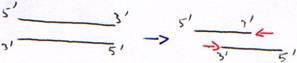
L’activité polymérase est dix fois plus rapide que l’activité exonucléase. Cette dernière sert donc à diminuer la quantité d’erreurs : c’est une activité de correction de la DNA polymérase.
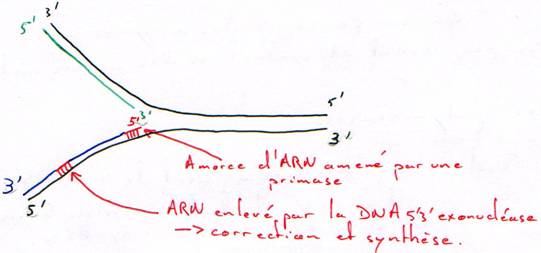
Activité 5’3’ exonucléase.

Cette activité est une activité de réparation.

Cette activité permet de réparer plus que ce qui est nécessaire : elle permet ainsi le renouvellement de l’ADN.
α La DNA polymérase I de E. coli.
Elle possède les trois activités précédentes. C’est une enzyme de 100 kDa, composée de deux fragments (un de 70 kDa et un autre de 30 kDa).
Le fragment qui possède les activités 5’3’ polymérase et 3’5’ exonucléase est appelé fragment de Klenow.
β Les DNA polymérases T4 et T7.
Ces enzymes ne possèdent que les activités 5’3’ polymérases et 3’5’ exonucléase. Quand la T7 est modifiée, elle n’a plus que l’activité polymérase.
γ La Taq DNA polymérase.
La Taq est la DNA polymérase de Thermus aquaticus (bactérie vivant à 80°C). Cette enzyme ne possède que la fonction polymérase. Toutefois elle peut synthétiser en continue et à haute température sans être dénaturée.
Application de la Taq :
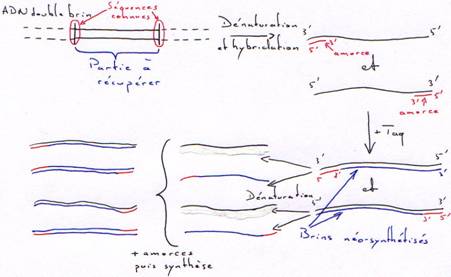
On obtient rapidement une amplification de la partie du brin comprise entre les amorces. Au bout du 40ème cycle et à partir d’un seul exemplaire, on obtient 1012 copies.
A partir d’un génome complet, on peut extraire une molécule bornée des deux côtés par les amorces et en obtenir un très grand nombre.
b Les DNA polymérases ARN dépendantes (les « reverse-transcriptases »).
On trouve ce type de polymérases chez les rétrovirus et dans les cellules eucaryotes mais en faible quantité.
Ces DNA polymérases ont une activité 5’3’ polymérase et nécessitent une amorce pour synthétiser ; elles ont souvent une activité de coupure de l’ARN quand il est hybridé (RNase H).
On purifie un ARNm (polyadénylé) par une hybridation avec un oligo-dT.
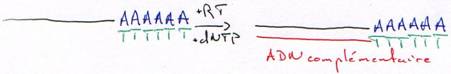
c DNA polymérase terminal-transférase.
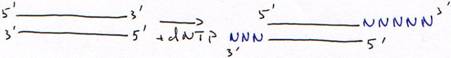
Ajout d’un seul nucléotide :
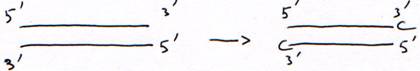
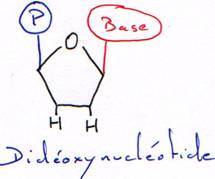
Avec un acide aminé sans fonction OH en 3’, la synthèse est arrêtée au bout d’un nucléotide. Cet acide aminé sans fonction OH est dit didéoxynucléotide.
B Les enzymes de dégradation.
1 Les RNA polymérases.
Les RNA polymérases agissent au niveau de la transcription. Elles synthétisent de 5’ vers 3’. Elles reconnaissent l’ADN double brin ainsi qu’un promoteur qu’il porte (site de reconnaissance et site de départ).

La transcription peut être dans les deux sens. Ce sens est définie par le promoteur.
2 Les DNAses.
Les DNAses sont des enzymes qui digèrent l’ADN. Il existe aussi des RNAses et des nucléases.
aDNase à reconnaissance non spécifique.
La DNase la plus connue est la DNase I qui reconnaît l’ADN double brin et qui le coupe (chez E. coli).
La nucléase S1 ne coupe que l’ADN simple brin.
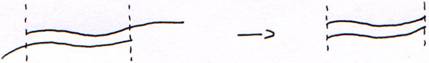

b Les DNAses de restriction.
Ce sont des enzymes qui digèrent l’ADN double brin sur des séquences spécifiques. Elles reconnaissent des palindromes.

Fragment reconnu par l’enzyme de restriction Eco R1 de E. coli.
Ces enzymes de restriction donnent deux types de coupures : des coupures franches (au même niveau sur les deux brins) et des coupures à extrémités sortantes.
3 Les RNAses.
Les RNAses vont couper l’ARN.
La RNase A : elle coupe le simple brin en 3’ des résidus pyrimidiques (après C et U) et donne des nucléotides 3’phosphate.
La RNase T1 : elle coupe du simple brin après G et donne un 3’phosphate.
La RNase H : chez les rétrovirus, elle élimine les brins d’ARN rétro-transcrits. Elle coupe après tous les nucléotides
4 Ligase.
La T4 DNA ligase fonctionne sur de l’ADN double brin avec au moins, une liaison OH-Phosphore.
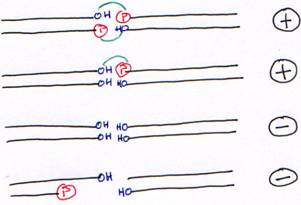
La T4 RNA ligase lie un ARN (du simple brin) avec son extrémité 5’ phosphatée sur une extrémité 3’ hydroxylée. On pourra mettre de l’ADN avec l’extrémité 5’ phosphatée.
5 Phosphatase, kinase et méthylase
Les phosphatases catalysent le retrait du groupement phosphate en 5’.
Les kinases avec la T4 polynucléotide-kinase : elle rajoute un phosphate en 5’.
Les méthylases vont particulièrement agir sur les sites de restriction en les méthylant pour empêcher l’action des enzymes de restriction.
II Les vecteurs.
Les vecteurs sont capables de transporter un fragment d’ADN et de l’amplifier par auto réplication.
A Les plasmides.
1 La transformation.
Il faut connaître les parties indispensables pour utiliser le plasmide comme vecteur.
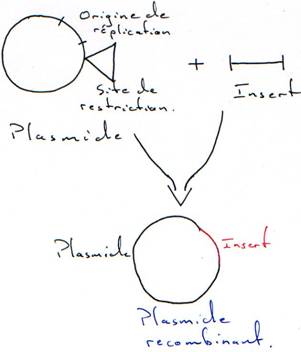
On a besoin d’un gène de résistance à un antibiotique (en général à l’ampicilline) pour pouvoir effectuer une sélection.
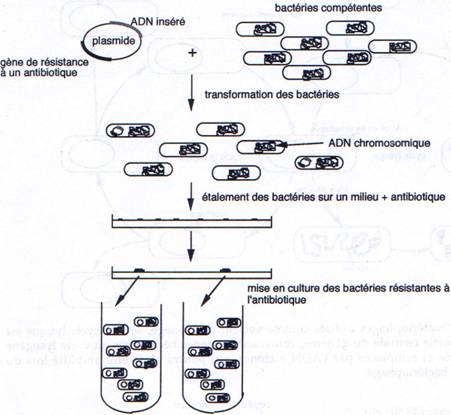
A la place d’avoir un seul site de restriction, on peut en avoir un second.
-GAATCC GAATCC- : c’est un polylinker.
On place, sur le plasmide, un gène qui code pour une enzyme qui colorera la bactérie.
L’intérêt d’un tel crible (coloré) est que l’on peut vérifier que le plasmide ne s’est pas religué seul.
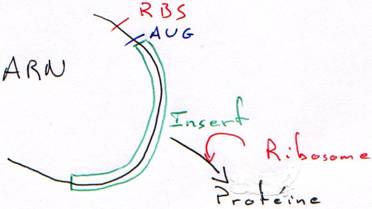
Quand on place un insert, le plasmide est recombinant. La RNA polymérase va pouvoir transcrire cet insert. Toutefois, ce dernier n’est en phase qu’une fois sur trois. Quand il n’est pas en phase, il n’y a pas de synthèse de protéine.
2 Les promoteurs des RNA polymérases.
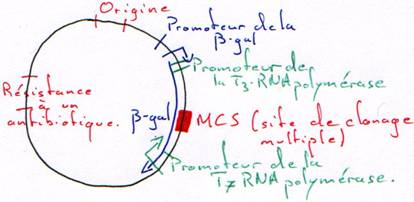
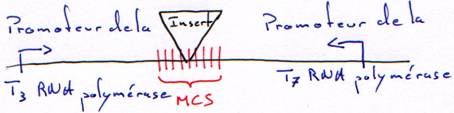
Le vecteur est un morceau d’ADN qui permet d’amplifier la synthèse de l’ADN à transcrire.
B Les phages.
Les phages sont des virus à bactéries dont on peut isoler la descendance.
1 Le phage λ.
Le phage l est un virus à ADN double brin, linéaire de 50 kb. Au milieu du génome, on trouve une partie de 20 kb qui n’est pas indispensable au cycle lytique. Elle peut être remplacée par un ADN étranger.
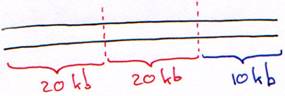

Le phage l est encapsidé, il reconnaît un récepteur au maltose, s’y fixe et injecte son ADN qui sera utilisé pour la transformation.
On est capable de fabriquer ce virus in vitro : protéines + ADN + encapsidation à virus
On peut aussi sélectionner un phage recombinant de phages non-recombinants.
Méthode de préparation du phage l :
On retire d’abord le fragment de 20 kb inutile et on obtient un bras droit de 20 kb et un bras gauche de 10 kb.
On sépare les deux fragments par centrifugation zonale.
On ajoute l’ADN recombinant que l’on va fixer par ligation.
Remarque : dans la capside, il ne rentre qu’un fragment d’ADN que 50 kb (+ ou – 5 kb).
Quand il rentre dans la bactérie, il devient circulaire avec extrémités auto-complémentaires. Sa réplication est comme celle d’un plasmide.
2 Le phage M13.
C’est un phage de 6 kb à ADN simple brin. Il rentre dans les cellules par les pili sexuels. Quand l’ADN est rentré, il passe en double brin puis commence à se répliquer comme un plasmide mais la réplication devient continue sur un seul brin : réplication en rolling circle. On obtient des ADN simples brins qui seront empaquetés et qui sortiront de la cellule.
Ce phage accepte de plus gros fragment d’ADN : on peut rajouter des sites de clonage multiple (MCS)
C Les systèmes hybrides.
1 Plasmide + origine de M13.
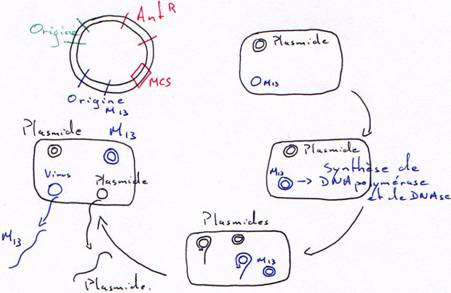
Ici, M13 est helper : il sert à produire les enzymes nécessaires à la synthèse de l’ADN simple brin.
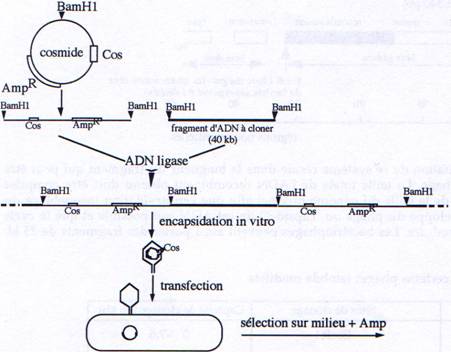
2 Les cosmides : plasmide + extrémités cos (cohésives) de λ.
Les cosmides sont des vecteurs hybrides portant à la fois :
- Des séquences d’origine phagique permettant leur encapsidation in vitro (séquence cos).
- Des séquences d’origine plasmidique : gène de résistance à un antibiotique et séquence permettant au plasmide de se répliquer dans une bactérie comme un simple plasmide (séquence ori).
Ceci permet de se dispenser des régions essentielles de l’ADN du phage et de construire des ADN recombinants portant jusqu’à 45 kb d’ADN étranger.
3 Les YAC (yeast artificial chromosome) ou chromosome de levure.
Ces vecteurs de clonages peuvent se présenter sous deux formes : une forme circulaire, permettant sa manipulation chez E. Coli (et en particulier l’insertion d’ADN exogène et son amplification è ils possèdent une séquence ori et un gène de résistance à un antibiotique) et une forme linéaire, obtenue après coupure par BamHI et EcoRI et qui permet d’obtenir les deux “bras” du YAC. Ces deux bras contiennent les télomères (TEL), une origine de réplication levure (ARS) un centromère (CEN) ainsi que des gènes (URA et TRP) utilisés pour la sélection des clones. La ligation de ces bras à un très grand fragment d’ADN humain (100 à 1000 kb) extrait de cellules en culture le transforme en pseudo chromosome de levure capable après introduction dans ce microorganisme de se répliquer (grâce à ARS), de stabiliser ses extrémités (grâce aux séquences TEL) et de se répartir entre les cellules filles lors de la division cellulaire (grâce à la séquence CEN).

III Les oligonucléotides.
On peut synthétiser des oligonucléotides jusqu’à 50 mères (ou bases). La synthèse se fait de 3’ vers 5’.
On greffe un nucléotide sur un support puis on rajoute un nucléotide protégé contre la fixation non-désirée d’un autre nucléotide à la fois. Entre chaque ajout d’un nucléotide, on enlève la protection. Finalement, on décroche l’oligonucléotide et obtient la séquence voulue ou variable.
A Le clonage.
Le génome humain comporte 3.109 paires de bases.
Pour en étudier une partie, on fait un tri que l’on va amplifier.
On fabrique des banques d’ADN génomique et des banques d’ARN. On transforme ces ARN en une population de clones. Une banque de cDNA est une banque génomique représentant l’ensemble du génome de la cellule de départ qui est découpé en fragments de taille connue.
Pour l’étude des ARNm, on dispose de deux techniques.
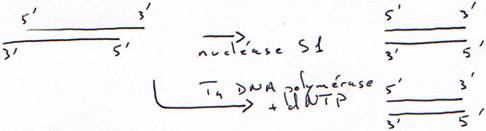
1 Technique à la nucléase S1.
On transforme l’ARN en ADN. On va donc hybrider cet ARN avec un oligo-dT.
Finalement, on obtiendra une copie qui n’est pas totale car elle aura perdu une partie son extrémité 5’ afin d’avoir des extrémités franches.
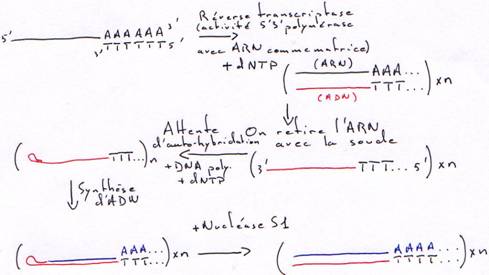
2 Technique à la RNase H.
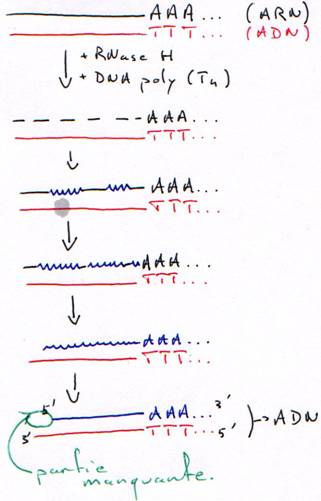
La DNA polymérase ne peut pas synthétiser le début car il n’y a pas d’amorce.
Dans ce cas aussi, il manquera un bout.
è Les cDNA ne sont pas complets : la séquence est aléatoire en fonction du lieu de la coupure.
B Utilisation des cDNA.
1 cDNA avec vecteur (et ligase).
La coupure est réalisée avec une enzyme de restriction (souvent avec Sau 3A) en digestion ménagée. On récupère alors des fragments dont la taille est proche de 20 kb.
Par centrifugation zonale (6 heures à 30 000 tours/minutes), les molécules sont séparées selon leur taille.
On réalise ensuite une électrophorèse pour vérifier la taille des fragments. On sélectionne ensuite les sections selon leur taille. On en trouve qui se recoupent car on utilise plusieurs génomes pour réaliser la digestion.
Les fragments sont ensuite insérés dans un vecteur qui est souvent le phage l. On garde son bras gauche de 10 kb et son bras droit de 20 kb. On rajoute l’insert de 20 kb, de la T4 DNA ligase et de l’ATP.
Le phage l ainsi modifié va être encapsidé si la taille du nouveau total est proche de 50 kb. Une fois que le phage a infecté les bactéries, on va réaliser un criblage de ces banques.
a Criblage avec antibiotiques.
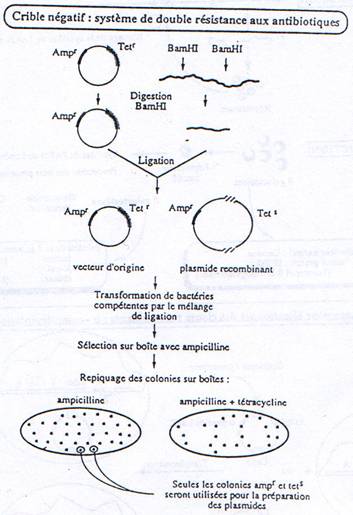
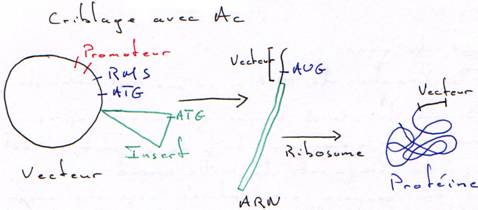
b Criblage Immunologique.
Tous les clones de la banque seront traduits. On sélectionnera les mutants qui fixeront l’anticorps correspondant à la protéine de l’insert.
2 Ajout d’un linker.
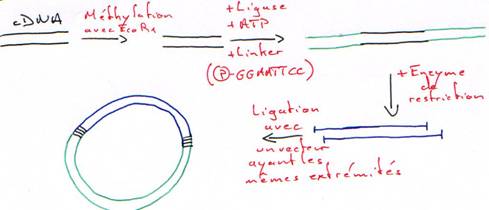
- Revenir en haut
- Aller à la partie 1 (Dénaturation et Hybridation)
- Aller à la partie 2 (Marquage des acides nucléiques)
- Aller à la partie 3 (Les outils du génie génétique)
- Aller à la partie 4 (Séquençage des acides nucléiques)
- Aller à la partie 5 (Mutagenèse dirigée)
Aucun commentaire jusqu'à présent.