Biologie Moléculaire :
Chapitre 3 : Réplication, Transcription et Traduction
Partie 1 : Réplication.
Télécharger le document (chapitre 3 entier) sur le serveur FTP ou sur le serveur web (HTTP).
I Généralités.
ALes expériences de Taylor (1957- incorporation de thymidine 3H) et de Meselson et Stahl (1958-incorporation de 15N).
Ces expériences ont permis de démontrer que la réplication s’effectue par un mécanisme semi-conservatif (les deux autres hypothèses impliquaient des modèles conservatif ou dispersif).
BEn 1960, par une expérience de Pulse-Chase en thymidine 3H…
…Cairns a montré que la réplication s’effectuait de manière bidirectionnelle à partir d’une (procaryotes) ou de plusieurs origines de réplication (eucaryotes : le nombre d’origines de réplications dépend de la taille du génome à répliquer – la phase S dure environ 2h – et peut atteindre plusieurs milliers). Chaque origine de réplication est donc constituée de deux fourches de réplication.
La réplication est une réaction rapide : 1000 nucl./sec/fourche de réplication chez les bactéries et 50 nucl./sec chez les mammifères. Chez ces derniers, la réplication s’accompagne de la synthèse protéique des histones qui s’assemblent pour former la structure chromatinienne. Cette étape supplémentaire ralentit la vitesse de polymérisation.
Enfin, la réplication est une réaction asymétrique qui s’effectue uniquement dans le sens 5’à3’. Cette asymétrie est liée à l’activité des ADNs polymérases :
- nécessité d’avoir une amorce ;
- le dernier résidu de l’amorce doit être apparié et avoir un groupement hydroxyle libre (3’OH) ;
- nécessité d’avoir une matrice ;
- le nucléotide triphosphate entrant apporte l’énergie nécessaire à la formation de la liaison phosphodiester.
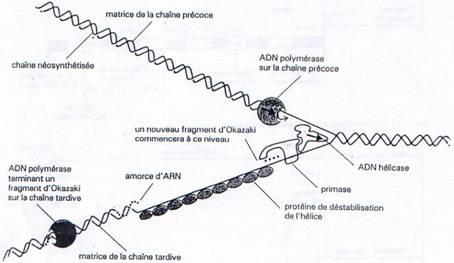
On distingue ainsi au niveau de chaque fourche de réplication, un brin précoce qui subit une synthèse continue et un brin tardif dont la synthèse s’effectue de manière discontinue dans un sens opposé à celui de la progression de la fourche de réplication.
II Mécanisme.
On distingue trois étapes au cours de la réplication : initiation, élongation, terminaison.
A L’initiation.
Une protéine d’initiation reconnaît une séquence particulière correspondant à l’origine de réplication.
Une ADN hélicase se fixe sur cette protéine d’initiation et va dérouler la double hélice en se déplaçant le long de la chaîne.
Des protéines vont stabiliser 1’ADN sous forme simple brin (SSB : Single Strand Binding protéine ou HDP : Helix Destabilizing Protéine)
Enfin, une ARN primase va se fixer au niveau de l’ADN hélicase et constituer ainsi le primosome. Cette enzyme va synthétiser de courtes séquences d’ARN (10 nucl.) qui vont par la suite servir d’amorce à l’ADN polymérase.
Une fois que l’ADN est localement sous forme simple brin (hélicase et SSB), et que la primase a synthétisé une amorce, l’ADN polymérase peut commencer la synthèse du brin complémentaire au brin parental. Au bout de quelques déoxyribonucléotides incorporés, l’initiation est terminée.
B L’élongation.
Pour que la fourche de réplication puisse continuer à avancer il faut dérouler la double hélice. Il faudrait que chaque double brin d’ADN parental (dans le cas d’un génome linéaire) tourne sur lui même à une vitesse d’environ 5 tours/sec chez un eucaryote et 100 tours /sec chez un procaryote. L’énergie ainsi libérée reviendrait à faire bouillir les cellules !!!
Une solution plus compatible avec la vie consiste à faire une coupure sur l’un des deux brins de la matrice, à laisser ce brin tourner autour de l’autre puis à le raccrocher ; c’est le rôle des topo isomérases de type I.
Dans le cas des génomes circulaires, c’est l’intervention de deux topo isomérases de type II (qui coupe les deux brins d’ADN) dont la gyrase qui va permettre de séparer les deux brins parentaux.
Ces problèmes de contraintes structurales réglés, l’ADN polymérase va pouvoir synthétiser en continu à partir d’une chaîne parentale un nouveau brin d’ADN, le brin précoce.
Par contre sur l’autre chaîne parentale, la synthèse s’effectue de manière discontinue. La primase va régulièrement synthétiser des amorces d’ARN à partir desquelles l’ADN polymérase va synthétiser les fragments d’Okasaki. Ces fragments sont reliés les uns aux autres par l’ADN ligase, après que l’ADN polymérase ait dégradé l’amorce ARN (grâce à son activité exonucléase 5’à 3’) puis re-synthétisé la partie manquante.
C La terminaison.
Dans le cas d’un ADN circulaire (procaryotes), la terminaison est réalisée lorsque les deux fourches de réplication se rencontrent (la topo isomérase de type IV assure l’étape de ligation).
Pour l’ADN linéaire, les fourches de réplication s’arrêtent soit lorsqu’elles rencontrent une autre fourche de réplication, soit lorsqu’elles arrivent à l’extrémité d’un chromosome.
Pour les génomes linéaires eucaryotes, une télomérase permet d’ajouter quelques nucléotides à l’extrémité du brin tardif, de manière à ne pas raccourcir les chromosomes à chaque réplication.
- Revenir en haut
- Aller à la partie 1 (Réplication)
- Aller à la partie 2 (Transcription)
- Aller à la partie 3 (Traduction)
j’aime bien comprendre le mecanisme detaillé de la replication d’adn pour les procaryote et aussi les eucaryotes et merci