BGU 02 :
Génétique, Chapitre 4, Régulation de l’expression génique.
Télécharger le document sur le serveur FTP ou sur le serveur web (HTTP).
I Expression génique.
L’ARN nucléotidique n’existe pas dans la cellule. Dès que l’ARN est synthétisé, il est pris en charge par les ribosomes pour éviter l’action des nucléases. Il y a donc un couplage transduction/traduction du fait que la bactérie n’a pas de noyau.
Si l’on a un opéron ABC, et qu’il y ait une mutation non-sens dans B, on n’aura, ni la protéine B, ni la protéine C, car le gène C sera digéré par la nucléase (l’effet polaire) et aussi car la transcription ne va pas jusqu’à C.
II Régulation.
Il y a plusieurs niveaux de régulation :
- Initiation de la transcription : activation ou non de promoteur.
- Post-transcriptionnel, qui joue sur la stabilité des messagers.
- Initiation de la traduction : accès ou non aux séquences reconnues par les ribosomes (Shine Dalgarno).
- Post–traductionnel : vitesse de traduction ou stabilité de la protéine.
Le niveau le plus économique est la régulation de l’initiation de la transcription.
A Initiation de la transcription.
On s’intéresse à la division cellulaire. On a cherché des mutants de division. On parle de gènes essentiels donc on cherche des mutants conditionnels : thermosensibles (pousse à 30°C et non à 42°C).
Parmi ceux qui ne poussent pas à 42°C on va chercher les mutants de division cellulaire. Lorsqu’on les fait pousser à 37°C, il y a deux cas :
- Les bactéries ne se sont pas divisées et sont sous forme de filament.
- Les bactéries se divisent bien.
On doit confirmer que se sont bien les filaments qui sont mutants de division cellulaire. On les remet alors à 30°C, puis à 42°C.
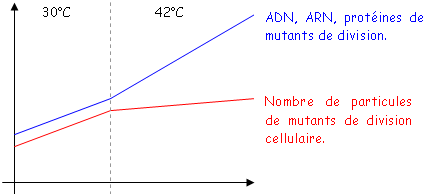
Le nombre de particules n’augmente plus car il n’y a plus de filamentation : il n’y a plus de division mais le métabolisme fonctionne toujours donc, les quantités d’ADN, d’ARN et de protéines croissent. On a isolé plusieurs mutants de division dont ftsZ84 (le gène ftsZ) code pour la protéine ftsZ qui polymérise pour former un anneau. La constriction de ftsZ nécessite le passage GTP –> GDP pour finalement cliver la cellule mère.
On a recherché des partenaires de ftsZ84 par recherche de suppresseurs de la mutation ftsZ84 qui sont exprimés à forte dose : ce sont les « suppresseurs multicopies ». On réalise la digestion par Sau3A d’ADN de E. coli pour obtenir des fragments d’environ 2,5Kb. On prend une autre souche portant un vecteur de clonage (un plasmide) portant une Ori, un gène de résistance à un antibiotique et un site de clonage multiple. On fait pousser avec l’antibiotique, on récupère le plasmide et on réalise une digestion complète avec BamH1 : les coupures sont les mêmes qu’avec Sau3A. On va mettre le plasmide digéré et de nombreux fragments d’ADN dans un eppendorf puis faire agir une ligase pour insertion : on a alors une banque génomique. Dans le mélange de ligation, on transforme ftsZ84 puis on étale sur milieu riche avec antibiotique (celui du départ) pour ne sélectionner que les transformants puis on incube à 42°C : C’est une double sélection.
En suite, on séquence l’insert et on l’injecte dans la banque de données et on retrouve à quoi correspond l’insert et quels sont les gènes qui suppriment la mutation ftsZ84. On va donc sortir ce gène ftsZ84 sauvage mais aussi, le gène rcsB qui code pour un régulateur transcriptionnel. RcsB supprime la mutation ftsZ84 en sur exprimant le gène ftsZ84. En effet, la quantité de protéines produites est plus importante que l’inactivation de ces protéines.
On va prouver ceci en utilisant les « fusions transcriptionnelles ».
Le gène rapporteur n’a plus son promoteur donc, il est transcrit par le promoteur ftsZ84. En présence de rcsB, on a 50 fois plus de produit du gène rapporteur, donc, rcsB active ftsZ.
Comment rcsB agit sur ftsZ ?On effectue une analyse par délétion : on va effectuer la délétion partielle du promoteur de ftsZ84 et doser le produit du gène rapporteur. On trouvera alors où est le site d’action de rcsB. Dans le cas d’une étude par analyse par mutation ponctuelle, on mute systématiquement chaque base. On peut aussi réaliser une analyse par transcription in vivo pour montrer que rcsB active directement la transcription. Pour cela, il faut une matrice ADN, RNApolymérase, des NTP marqués et du tampon pour pouvoir synthétiser de l’ARN. Ensuite, on fait migrer les produits obtenus sur un gel d’électrophorèse en conditions dénaturantes. En présence de rcs (ou rcs+), on voit une beaucoup plus forte activité transcriptionnelle de ftsZ. Rcs est suffisant pour activer la transcription de ftsZ.
RcsB est un régulateur transcriptionnel. Il appartient à un système à deux compartiments : deux gènes rcsB et rcsC. Ici, le premier gène (C) code pour le « senseur » et le second (B) code pour le régulateur.
Le gène rcsC code pour une protéine transmembranaire qui « ressent » un signal extérieur abîmant la membrane. Il y a alors auto-phosphorylation de la partie cytoplasmique de rcsC. Le phosphate est ensuite transmit à rcsB qui est alors seulement activé. Elle va avoir une action sur une cible : dans ce cas, ftsZ.
On sait que rcsC/rcsB régule : la division cellulaire, la réplication de l’ADN (en l’arrêtant), la synthèse de la capsule bactérienne (en l’activant), la synthèse des flagelles pour la mobilité et le chimiotactisme, le système de résistance à des drogues multiples. Le complexe rcsC/rcsB va intervenir dans la régulation de plus de 50 gènes : c’est un régulateur global de bactérie. Le fait que rcsB se fixe avant la séquence –35 fait que la fixation de la polymérase est augmentée par rcsB ? Augmentation de la transcription. Si rcsB se fixe sur la séquence –3, il empêche la fixation de la polymérase et réprime donc la transcription.
Aucun commentaire jusqu'à présent.